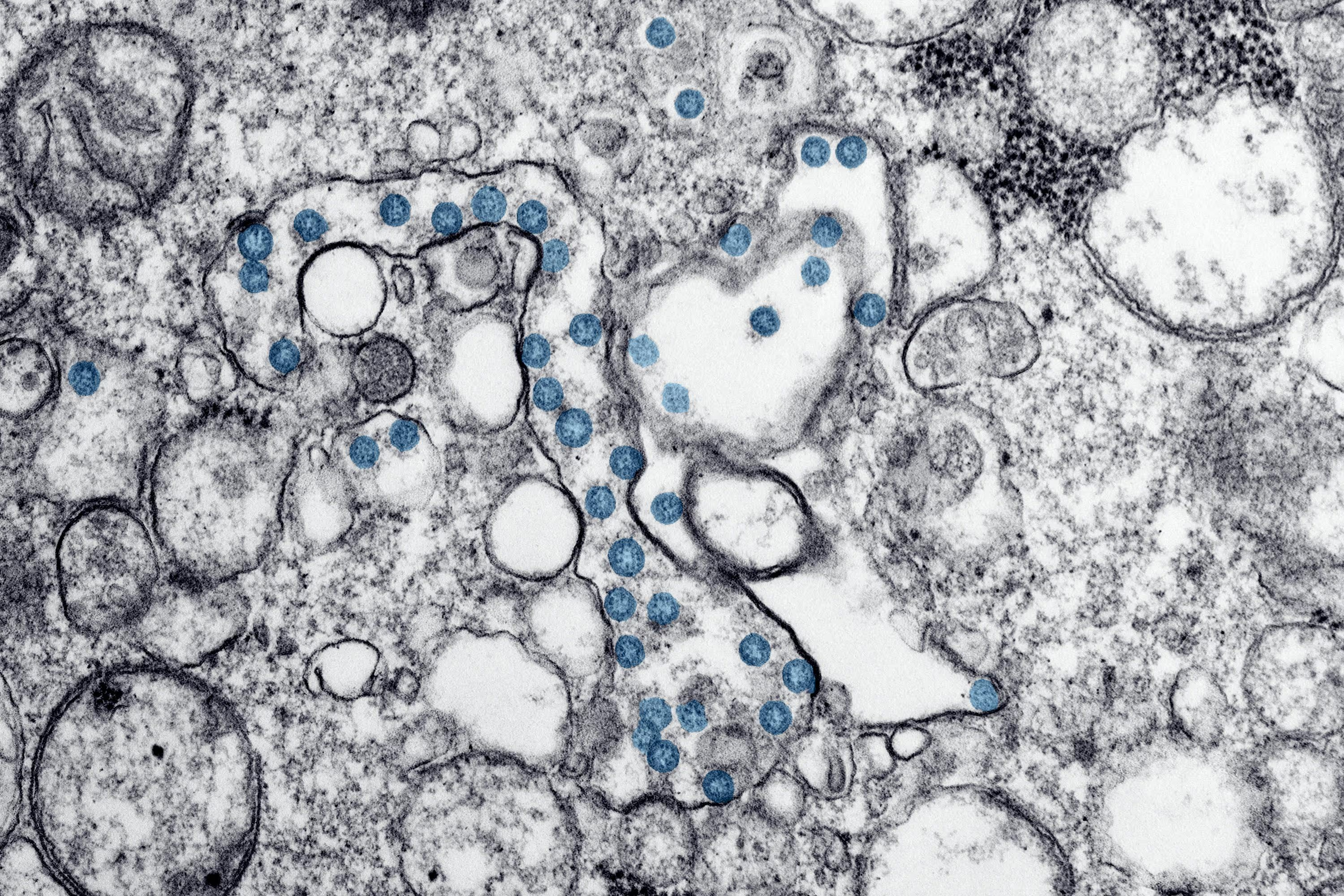
Einführung: Warum die Segmentierung von Tumoren mit mehreren Klassen für KI im Gesundheitswesen von entscheidender Bedeutung ist
In den letzten Jahren hat sich künstliche Intelligenz (KI) zu einer transformativen Kraft in der Onkologie entwickelt, die eine frühere Krebserkennung, eine schnellere Diagnose und eine individuellere Behandlung ermöglicht. Im Mittelpunkt dieser Fortschritte steht eine wichtige Aufgabe: Tumorsegmentierung.
Medizinische Daten aus der realen Welt sind jedoch komplex. Tumoren existieren selten isoliert. Ein einzelner Scan kann Folgendes enthalten mehrere Tumorarten, Noten, Formen, und Gewebestrukturen— alles anspruchsvoll Mehrklassensegmentierung für genaues Modellieren.
Im Gegensatz zur binären Segmentierung, bei der Pixel als Tumor oder Nicht-Tumor klassifiziert werden, Multiklassen-Tumorsegmentierung baut verschiedene Tumor-Subregionen ab, wie zum Beispiel:
- Tumorkern
- Nekrotischer Bereich
- Ödem
- Infiltrierender Rand
- Zystische Komponenten
- Spezifische Tumorsubtypen (z. B. Glioblastom vs. Oligodendrogliom)
Dieser Artikel dient als vollständige Anleitung zur Navigation im Herausforderungen, Arbeitsabläufe für Anmerkungen, Werkzeuge, und KI-Strategien für die Segmentierung von Tumoren mit mehreren Klassen. Ganz gleich, ob Sie ein klinisches Modell erstellen oder WSIs in der Pathologie mit Anmerkungen versehen, diese Inhalte helfen Ihnen dabei, skalierbare, präzise Arbeitsabläufe zu entwerfen, die den realen Anforderungen entsprechen.
1. Was ist Multiklassen-Tumorsegmentierung?
💡 Definition
Die Segmentierung mehrerer Tumoren ist der Prozess der Markierung mehrerer tumorbezogener Regionen in einem medizinischen Bild — jede mit einem eindeutiges Klassenlabel. Anstatt eine einzige binäre Maske zu erzeugen, gibt das KI-Modell aus eine eigene Klasse für jede Region.
Eine segmentierte Gehirn-MRT könnte beispielsweise Folgendes beinhalten:
- Klasse 0: Hintergrund
- Klasse 1: Tumorverstärkender Kern
- Klasse 2: Ödem
- Klasse 3: Nekrotisches Gewebe
🧠 Warum es wichtig ist
- Bietet eine granulares Verständnis der Tumorstruktur
- Aktiviert volumenbasierte Behandlungsplanung (z. B. Strahlungs-Targeting)
- unterstützt Einstufung von Tumoren und Überwachung der Progression
- Nützlich in multimodale Bildgebung (CT, MRT, PET) wo verschiedene Komponenten unterschiedlich reagieren
📚 Erfahren Sie, wie die Mehrklassensegmentierung die Überwachung von Gehirntumoren ermöglichte, in der BRATS-Herausforderung
2. Bildgebungsmodalitäten, die bei der Mehrklassensegmentierung verwendet werden
Die multiklassenübergreifende Tumorsegmentierung umfasst mehrere Bildgebungsarten:
🧠 MRT (Magnetresonanztomographie)
Häufig bei Weichteiltumoren (z. B. Gliome, Prostatakrebs). Verschiedene Sequenzen (T1, T2, FLAIR) enthüllen Tumor-Subregionen.
CT (Computertomographie)
Wird bei Lungen-, Leber- und Darmtumoren verwendet. Weniger Kontrast als MRT, aber allgemein zugänglich.
🧫 Histopathologie (WSIs)
Wird bei Brust-, Haut- und Prostatakrebs eingesetzt. Ermöglicht die hochauflösende Segmentierung von Tumorkomponenten auf Zellebene.
🔬 HAUSTIER-CT/PET-MRT
Fügt Stoffwechselinformationen hinzu, die für die Definition aktiver Tumorregionen entscheidend sind (z. B. FDG-Aufnahme bei Lymphomen).
🧾 Einen Open-Source-MRT-Datensatz mit Multiklassen-Tumorsegmentierung finden Sie unter BRATs 2021
3. Häufige Herausforderungen bei der Segmentierung von Tumoren mit mehreren Klassen
Trotz ihrer Leistungsfähigkeit bietet die Mehrklassensegmentierung mehrere technische, medizinische und betriebliche Herausforderungen:
⚠️ 3.1. Ungleichgewicht zwischen den Klassen
Tumorklassen wie Nekrose können < 5% des Bildvolumens einnehmen, während Ödeme dominieren können. Dieses Ungleichgewicht kann das Lernen verzerren.
Lösung: Nutze Verlustfunktionen wie Würfelverlust oder Fokusverlust, um unterrepräsentierten Klassen mehr Gewicht zu geben.
🧩 3.2. Visuelle Ähnlichkeit zwischen den Klassen
Subregionen wie Ödeme und sich nicht verstärkende Tumoren können nahezu identisch erscheinen, insbesondere bei Scans mit niedriger Auflösung.
Lösung: Kommentieren Sie mit mehreren Bildgebungsmodalitäten oder Sequenzen (z. B. T2 für Ödem, T1-Gd für eine Verstärkung).
Bild 3.3. Mehrdeutigkeit der Anmerkung
Selbst erfahrene Radiologen und Pathologen sind sich möglicherweise nicht einig, wo eine Klasse endet und eine andere beginnt.
Lösung: Erstellen Sie eine QA-Schleife mit einer Entscheidung oder berechnen Sie eine Vereinbarung zwischen den Annotatoren (z. B. Cohens Kappa).
🧠 3.4. Ausrichtung auf mehrere Modalitäten
MRT, CT und PET können falsch ausgerichtet sein, was zu Klasseninkongruenzen zwischen den Bildern führen kann.
Lösung: Verwenden Sie Registrierungstools wie ANTs oder ITK, um die Modalitäten vor der Annotation abzugleichen.
🖼️ 3,5. Datenmenge und Verarbeitung
Gigapixel-Pathologiefolien und große volumetrische Scans erfordern Kachelung, Patch-Extraktion und Hardwarebeschleunigung.
Lösung: Verwenden Sie kachelbasierte Annotationsplattformen und hybride CPU/GPU-Pipelines.
📚 Das MIT-Papier erörtert das Klassenungleichgewicht bei der Tumorsegmentierung und wie synthetische Augmentation hilft.
4. Annotationsstrategien für die Segmentierung von Tumoren mit mehreren Klassen
Präzision beginnt bei der Etikettierung. So gehen Experten an komplexe, mehrklassige Tumorannotationen heran:
🖊️ 4.1. Polygonale Anmerkung
Wird verwendet, um unregelmäßige Tumorformen zu skizzieren — besonders hilfreich für histologische oder dermatopathologische Bilder.
- Ideal für nicht rechteckige Tumorgrenzen
- Arbeitsintensiv, aber hochgenau
- Unterstützt von Tools wie QuPath, Aiforia und Labelbox
🟪 4.2. Semantische Segmentierung (pixelweise Masken)
Jedes Pixel ist mit einer Klasse beschriftet, weshalb sich diese Methode ideal für das Training von CNNs mit volumetrischen Daten eignet.
- Häufig bei MRT- und CT-Schnitten
- In der Regel mithilfe halbautomatisierter Workflows
- Maskieren Sie Arrays, die in Formaten wie gespeichert sind
.npy,.png, oder DICOM-SEG
🧩 4.3. Volumetrische 3D-Segmentierung
Besonders relevant für Gehirn-, Leber- und Lungenkrebs. Annotatoren arbeiten segmentübergreifend, um die Klassenkonsistenz im 3D-Raum zu gewährleisten.
- Ermöglicht eine genaue Schätzung des Tumorvolumens
- Wird auf Plattformen wie ITK-SNAP oder 3D Slicer verwendet
- Exportiert im NIFTI- oder DICOM-Format
📍 4,4. Segmentierung von Instanzen
Jede Tumorregion ist segmentiert und separat beschriftet — auch wenn es sich um dieselbe Klasse handelt. Entscheidend für die Unterscheidung zwischen multifokale Tumoren.
🔁 4,5. Anmerkung zum Modell in der Schleife
KI liefert erste Segmentierungen, die von Menschen korrigiert werden. Ideal, um große Datensätze mit mehreren Klassen zu beschleunigen.
- Feedback-Schleife verbessert zukünftige Prognosen
- Wird in Tools wie Encord Active oder Supervisely Auto-Labeling verwendet
- Reduziert den Zeitaufwand bei komplexen Datensätzen um bis zu 60%
📘 Lies das Nature zur Model-in-Loop-Annotation in Brustkrebs-KI.
5. Tools und Plattformen zur Unterstützung der Markierung von Tumoren mit mehreren Klassen
Hier finden Sie praxiserprobte Tools, die mehrstufige Segmentierungsworkflows für alle Arten der medizinischen Bildgebung unterstützen:
💻 Tool-Beispiele:
- 3D-Hobel — Open Source; ideal für 3D-MRT/CT-Tumor-Annotationen
- ITK-SNAP — Intuitive GUI für medizinische 3D-Bildsegmentierung
- QuPath — WSI-fokussiert, wird in der Histopathologie eingesetzt
- Labelbox//Encord — Unternehmenstauglich mit KI-gestützter Etikettierung
- MONAI-Etikett — KI-Modell-in-the-Loop-Annotation in Echtzeit
🔗 DICOM-native Tools finden Sie in der OHIF-Viewer wird in Forschungskrankenhäusern weltweit eingesetzt.
6. KI-Modellarchitekturen für die Segmentierung von Tumoren mit mehreren Klassen
Um eine Segmentierung mit mehreren Klassen durchzuführen, benötigen Sie Modelle, die dazu in der Lage sind Ausgabe mehrerer Etiketten. Hier sind einige beliebte Architekturen:
🔬 6.1. U-Net und Varianten
- U-Net, Achtung U-Net, U-Net++
- Ideal für biomedizinische Segmentierung (2D und 3D)
🧠 6,2. DeepLab V3+
- Nutzt Atrouswindungen für die Segmentierung auf mehreren Skalen
- Gute Ergebnisse bei der Vorhersage von Gewebearten mit mehreren Klassen
🧬 6.3. NNU-Netz
- Selbstkonfigurierendes Framework; war ein Top-Performer bei BRATs- und KITs-Wettbewerben
- Hervorragend für die Segmentierung von Gehirn- und Nierentumoren
📦 6.4. Transformatoren für die Segmentierung
- Modelle wie SEG Former oder Transunet bieten weitreichende Abhängigkeiten
- Nützlich in der Histopathologie, wo es auf eine feinkörnige Textur ankommt
🧪 Schauen Sie sich MONAI an für PyTORCH-basierte Tumorsegmentierungspipelines.
7. Anwendungsfälle in Klinik und Forschung
🧠 7,1. Gehirntumor (Gliom, GBM)
- Segmentierung des Kerns, Ödem, Nekrose
- Verfolgt die Tumorprogression im Laufe der Zeit
- Wird bei BRATs- und FETs-Wettbewerben verwendet
Höhe 7.2. Prostatakrebs
- Gleason-Sortierung mit mehreren Klassen
- Kernsegmentierung im Vergleich zur Peripherie
- Wird in KI-gestützten Pathologietools verwendet
7.3. Lungenkrebs
- Unterscheidet die Opazität von festem, halbfestem und gemahlenem Glas
- Verfolgt die Reaktion auf gezielte Therapien
🧫 7,4. Brustkrebs
- IDC- und DCIS-Klassifizierung
- Tumor vs. Stroma vs. Immunsegmentierung
- Entscheidend für HER2- und ER-Testworkflows
🔬 7,5. Leber- und Pankreastumoren
- Mehrphasige CT-Segmentierung
- Analyse der portalvenösen vs. arteriellen Phase
📖 TCGA und TCIA sind hervorragende Quellen für annotierte Multiklassen-Tumordaten.
8. Überlegungen zur Qualitätssicherung und Einhaltung der Vorschriften
- Konsensus-Annotation verwenden von mehreren Spezialisten
- Verfolgen Sie die Vereinbarung zwischen den Kommentatoren (Cohens Kappa, Dice Score)
- Stellen Sie die Anonymisierung sicher per HIPAA-Richtlinien
- Halten Sie sich an die DSGVO für alle in der EU ansässigen Patientenbildgebung
✅ Integrieren Sie automatische QA-Checks in Ihre Pipeline, um Fehler bei der manuellen Überprüfung zu reduzieren.
9. Trends und die Zukunft der multiklassenübergreifenden Tumorsegmentierung
🌐 Föderiertes Lernen
Krankenhäuser trainieren KI-Modelle anhand lokaler, mehrstufiger Tumordaten ohne Datenübertragung — und schützen so die Privatsphäre der Patienten.
🧪 Synthetische Daten
GANs und Diffusionsmodelle werden verwendet, um synthetische Tumorproben für unterrepräsentierte Klassen zu erstellen.
🧠 Schwache Aufsicht
Verwendet grobe Anmerkungen oder Tags auf Folienebene, um Segmentierungen auf Pixelebene zu generieren und so die Kosten für Anmerkungen zu reduzieren.
🧩 Fleckenübertragung und Normalisierung
Stellt sicher, dass die KI auf histopathologischen Objektträgern mit unterschiedlichen Farbprofilen generalisiert.
🔬 Erkunden Herausforderung bei der Fed-Tumorsegmentierung um föderiertes Lernen in Aktion zu erleben.
📌 Arbeiten Sie mit Experten für Multi-Class-Tumorsegmentierung zusammen
Benötigen Sie Hilfe beim Erstellen oder Kommentieren von Datensätzen zur Tumorsegmentierung mit mehreren Klassen?
Bei DataVLab, wir spezialisieren uns auf:
- Tumorannotation in Radiologie und Pathologie
- Model-in-Loop-Segmentierungsworkflows
- Volumetrische 3D-Etikettierung und Qualitätssicherung
- Konforme, von Experten geleitete Datenpipelines
📩 Kontaktiere uns um Ihre medizinische KI-Roadmap mit von Experten kommentierten, leistungsstarken Tumordaten zu beschleunigen.